Particelle di virus dell'influenza suina. Crediti: NIAID/Flickr. Licenza: CC BY 2.0
Come ogni anno, con il sopraggiungere della stagione invernale saremo esposti ai virus influenzali che causeranno problemi di salute e significative perdite economiche. In una piccola ma non trascurabile percentuale, ci saranno complicazioni e potenzialmente anche un aumento della mortalità tra neonati e anziani provocata dagli stessi virus. Ritorna perciò il tema della vaccinazione anti influenzale annuale. Da un’analisi del 2017 dei Centers for Disease Control and Prevention (CDC) di Atlanta, emerge che negli ultimi dieci anni l’efficacia della vaccinazione annuale è oscillata tra il 10 e 60 per cento. Una delle cause di questa efficacia variabile e non ottimale è la diversità dei virus influenzali circolanti rispetto a quelli utilizzati per la formulazione del vaccino, che viene aggiornata ogni anno con alcuni mesi di anticipo. Come riesce il virus a mantenersi all'interno della popolazione un anno dopo l'altro? Il nostro studio ha individuato quattro mutazioni che potrebbero avere un ruolo in questo processo, e che potrebbero suggerire una strategia da includere nello sviluppo di un vaccino universale contro l’influenza.
Trucchi virali per aggirare il sistema immunitario
I virus influenzali cambiano in continuazione attraverso due meccanismi definiti antigenic shift e antigenic drift. Il primo dipende dalla caratteristica peculiare del virus dell’influenza di essere costituito da otto segmenti di RNA indipendenti tra loro. Se una cellula aviaria e/o suina è infettata contemporaneamente con virus influenzali diversi, esiste la possibilità che si possa generare un virus di nuova composizione costituito da segmenti provenienti da virus diversi. Se il nuovo virus è in grado di compiere il salto di specie nell’uomo, questo non avrà memoria immunologica protettiva (ovvero anticorpi capaci di neutralizzare il virus) e il nuovo virus potrà diffondersi globalmente nel pianeta, spesso con morbilità e mortalità superiori al virus stagionale. Per fortuna, questo evento è raro; negli ultimi cento anni si è verificato quattro volte: nel 1918 con la famosa spagnola (con 500 milioni di persone infettate e 50-100 milioni di morti), seguita dall’asiatica nel 1958, l'Hong Kong nel 1968 e la suina nel 2009 (con mortalità significativamente inferiori).
Tuttavia, dopo le pandemie i virus influenzali non spariscono, ma si adattano e persistono nella popolazione attraverso l’acquisizione di mutazioni (antigenic drift) generate da errori di replicazione e pressione della risposta immunitaria. In questo modo, i virus pandemici progressivamente si trasformano in virus stagionali caratterizzati da una perdita della loro capacità patogenetica nell’uomo. Oltre alla classica risposta immunitaria all’infezione o al vaccino, basata sulla produzione di anticorpi neutralizzanti, le nostre cellule percepiscono la presenza di un virus grazie a sensori cellulari che riconoscono gli acidi nucleici (DNA o RNA) o le proteine virali. La conseguenza è la sintesi e attivazione di un insieme di proteine di risposta antivirale generalmente dipendenti dalla produzione di interferone da parte delle cellule infettate, una risposta corale che viene definita “innata” per distinguerla dal quella adattiva legata alla produzione di anticorpi e di cellule citotossiche specifici per l’agente infettante che emerge successivamente alla risposta immunitaria innata e anche grazie a essa.
TRIM 22, l'etichettatrice contro i virus
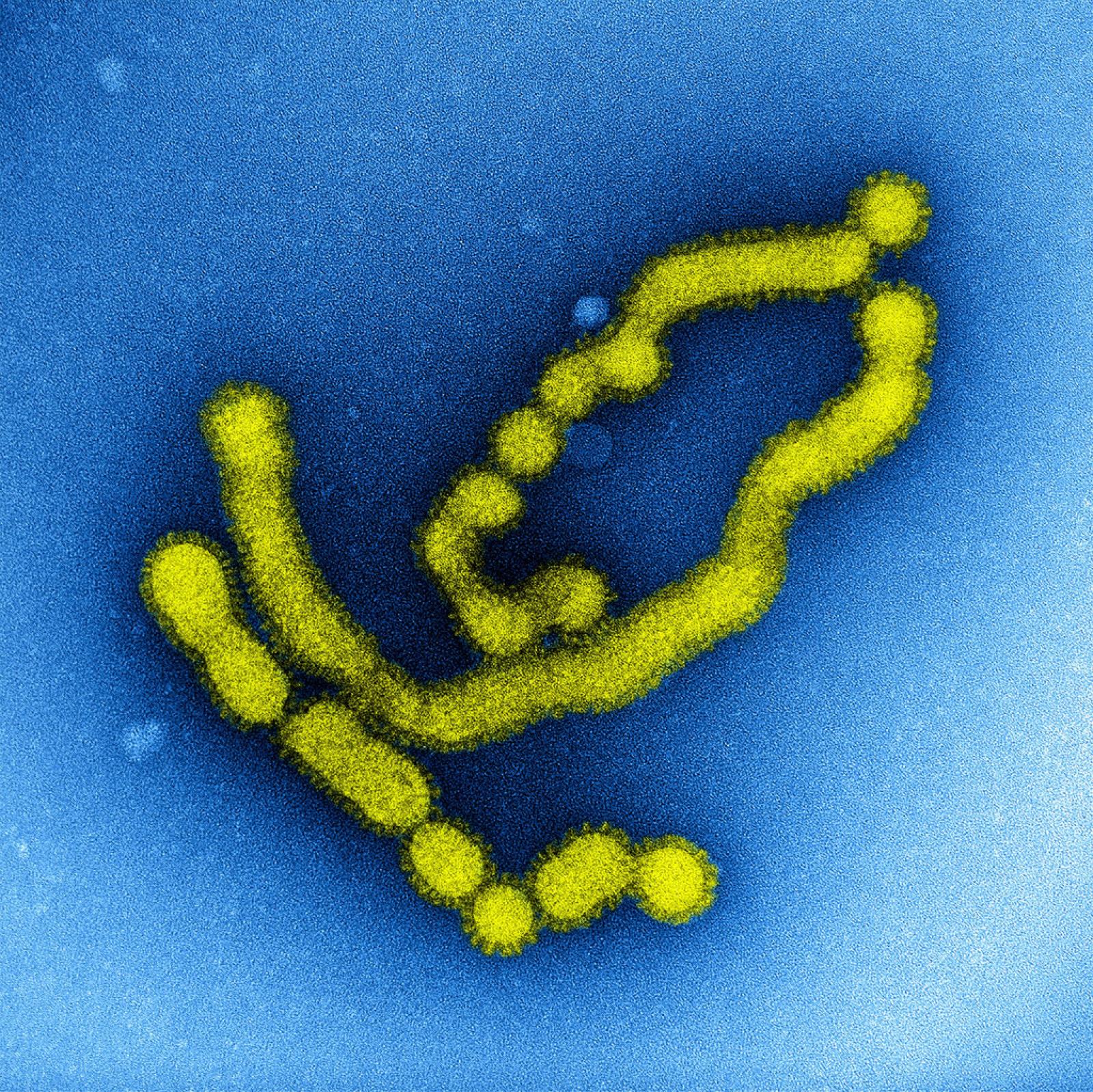
Tra le proteine di risposta antivirale indotta da interferone, abbiamo identificato da tempo TRIM22, una molecola in grado d’inibire l’infezione dei virus stagionali attraverso l’interazione con la nucleoproteina virale che avvolge come un rocchetto gli RNA della particella virale. Una volta legata alla nucleoproteina, TRIM22 le “attacca” molecole di ubiquitina, una sorta di etichetta che segnala al macchinario intracellulare deputato alla degradazione delle proteine (il proteasoma) che dev’essere eliminata (Figura 1). TRIM22 quindi induce la degradazione di una componente del virus indispensabile per la sua replicazione.
Quando abbiamo confrontato un virus pandemico a virus stagionali abbiamo osservato che il primo non era inibito da questo meccanismo. Per cercare di spiegare questo fenomeno siamo andati a confrontare le sequenze della nucleoproteina dei virus stagionali e di quelli pandemici osservando importanti differenze: al posto delle lisine, nei virus pandemici sono presenti quattro arginine, un altro amminoacido. Poiché l'"etichettatura" delle proteine spiegata sopra avviene solo sulle lisine (Figura 1), ci siamo chiesti quante e quali lisine della nucleoproteina fossero bersaglio di TRIM22.
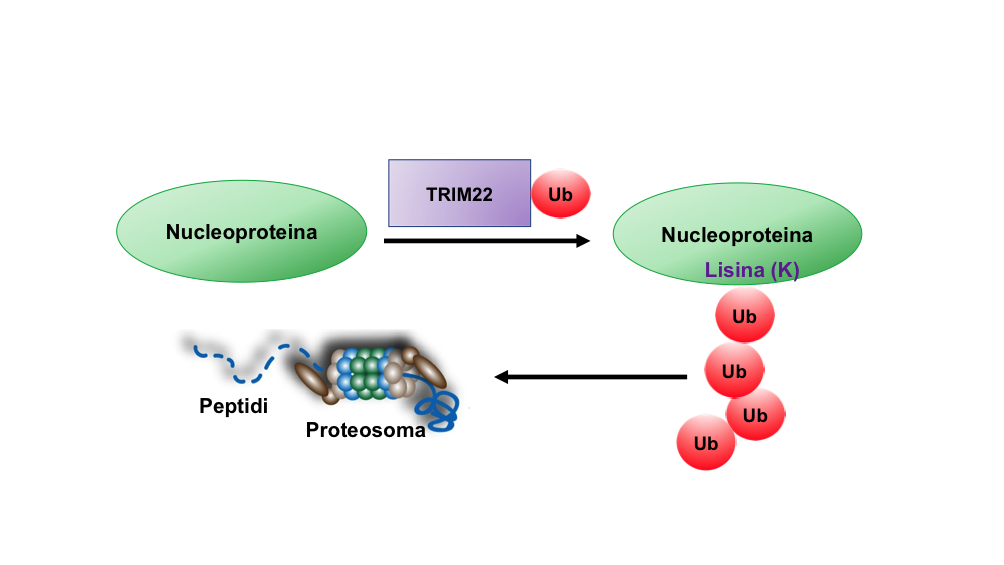
Figura 1. TRIM22 lega le molecole di ubiquitina alla nucleproteina dei virus influenzali stagionali utilizzando il residuo aminoacidico lisina (K). La “catenella” di ubiquitina guida la nucleoproteina al proteosoma, il macchinario intracellulare deputato alla degradazione delle proteine inclusa la nucleoproteina. Immagine di Elisa Vicenzi.
Cambia una lettera, cambia la parola
L’analisi cronologica delle sequenze virali dall'anno della pandemia spagnola (1918) alla pandemia suina del 2009 ci ha permesso di identificare un progressivo accumulo di queste mutazioni (Figura 2A). Il primo cambiamento da arginina a lisina si osserva nel 1936 nell’arginina in posizione 98, seguito da una seconda mutazione in posizione 422 nel 1940. Il terzo cambiamento avviene nel 1948 in posizione 293 e l’ultimo si verifica sull’aminoacido in posizione 446 nel 1977. Sorprendentemente, l’insorgenza della pandemia suina nel 2009, ha riportato le 4 arginine come nel 1918. Questi cambiamenti hanno una curiosa analogia con un gioco della Settimana Enigmistica: partendo da una parola (ossia una proteina virale), si effettua un cambiamento (mutazione) di una lettera (aminoacido) per volta, facendo attenzione che tutti gli intermedi abbiano un significato (ossia che tutti i virus fossero in grado di replicarsi), per ottenere infine una parola (proteina) completamente diversa. Nell’esempio, in quattro passaggi si arriva da “CASO” a “ROMA” (Figura 2B).
La nostra ricerca ha dimostrato un meccanismo molto simile: dalle quattro arginine presenti nel virus pandemico resistente a TRIM22 (Figura 2A), il virus si è evoluto cambiando un amminoacido per volta, fino ad accumulare, nell'arco di novant'anni, le quattro lisine del virus influenzale stagionale, suscettibile all’azione di TRIM22.
Figura 2. Un secolo di circolazione dei virus influenzali provoca il cambiamento di quattro residui di aminoacidi critici per la risposta a TRIM22. A) Quattro pandemie si sono succedute nel corso del XX e XXI secolo. Le mutazioni arginina-lisina sono indicate in ordine cronologico di comparsa. B) Analogia dell’evoluzione della nucleoproteina con un gioco della Settimana Enigmistica. Figura di Elisa Vicenzi con contributo di Eufemia Putortì.
Per poter dimostrare sperimentalmente che l’effetto di resistenza o suscettibilità all’effetto di TRIM22 fosse spiegato esclusivamente dai quattro cambiamenti arginina-lisina della nuceloproteina, abbiamo utilizzato la tecnica definita reverse genetics. Abbiamo creato molecole di nuceloproteina mutate e abbiamo osservato che solo il “mutante quadruplo”, cioè il virus con tutte e quattro le lisine mutate in arginine non era più suscettibile all’inibizione indotta da TRIM22, mentre la sua proteina non era più suscettibile all’attività di "etichettatura" di TRIM22. Abbiamo anche compiuto l’esperimento opposto, correggendo le arginine del virus pandemico con le lisine di quello stagionale e, nuovamente, il mutante quadruplo ha conferito suscettibilità all’inibizione da TRIM22.
Ragionando in termini vaccinali, vi sono due risposte immunitarie specifiche rilevanti per il virus influenzale. La prima è la produzione di anticorpi che riconoscono la proteina di superficie emagglutinina e che sono quindi in grado di neutralizzare il virus prima che infetti le cellule. La seconda risposta immunitaria specifica è basata sulla generazione di linfociti T citotossici che riconoscono e uccidono le cellule infettate che esprimono alla loro superficie frammenti peptidici di nucleoproteina (associati agli antigeni di istocompatibilità), sebbene questa risposta sia meno sfruttata in chiave vaccinale. Curiosamente, l’evoluzione del virus da pandemico a stagionale ha introdotto mutazioni nella nucleoproteina che la rendono suscettibile all’azione degradativa di TRIM22 nel proteasoma. Possiamo quindi ipotizzare che i peptidi generati dal proteasoma come conseguenza dell’effetto antivirale di TRIM22 possano anche stimolare la risposta immunitaria dei linfociti T citotossici finalizzata a eliminare le cellule infettate.
Questo è un aspetto contro-intuitivo: che vantaggio ha il virus a mutare, se questo lo rende più sensibile alla risposta immunitaria dell'ospite? In realtà, possiamo ipotizzare che queste mutazioni (e la conseguente suscettibilità a TRIM22), rientrino tra i meccanismi che determinano una attenuazione di patogenicità, ossia rendono il virus meno aggressivo. In termini evolutivi, un virus meno aggressivo può infettare un numero di persone maggiore rispetto a uno molto aggressivo. È proprio ciò che succede con l'influenza, che, a differenza di altri virus come ad esempio SARS o Zika, si ripresenta un anno dopo l'altro. Le mutazioni che abbiamo studiato potrebbero quindi rientrare in questo meccanismo di do ut des, per il quale il virus diventa sì suscettibile all'azione di alcune componenti del sistema immunitario, ma la ridotta patogenicità che questo comporta gli permette di persistere negli anni all'interno della popolazione ospite.
Inoltre, la ricerca per un vaccino influenzale universale è concentrata sulla risposta anticorpale. Ma se l'azione di TRIM22 permette di aiutare l’attivazione dei linfociti T citotossici, si potrebbe pensare di includere anche questi ultimi.
Bibliografia:
Pagani I, Di Pietro A, Oteiza A, Ghitti M, Mechti N, Naffakh N, Vicenzi E. Mutations Conferring Increased Sensitivity to Tripartite Motif 22 Restriction Accumulated Progressively in the Nucleoprotein of Seasonal Influenza A (H1N1) Viruses between 1918 and 2009. mSphere. 2018 Apr 4;3(2). pii: e00110-18. doi: 10.1128/mSphere.00110-18.