Il Nobel per la Chimica 2009, attribuito congiuntamente a Thomas A. Steitz (USA), Ada E. Yonath (Israele) e a Venkatraman (Venki) Ramakrishnan (Regno Unito), corona un'impresa scientifica eccezionale: la decifrazione a livello atomico della struttura dei ribosomi, le "fabbriche" cellulari delle proteine.
I ribosomi sono grandi aggregati macromolecolari costituiti da "grandi molecole" come RNA e proteine. La funzione dei ribosomi è polimerizzare, ovvere legare gli uni agli altri in lunghe catene, gli aminoacidi nell'ordine dettato dal messaggio genetico, "traducendo" appunto in catene polipeptidiche l'informazione contenuta nel DNA (Figura 1). Tutti i ribosomi sono composti da due parti di dimensioni disuguali, le subunità maggiore e minore, che hanno compiti distinti nella traduzione. La subunità minore lega l'RNA messaggero (il meteriale genetico che trasferisce l'informazione dal Dna alle proteine), individua il punto di partenza per la lettura del messaggio genetico e controlla che vengano scelti esattamente gli aminoacidi da esso specificati. La subunità maggiore fornisce l'attività catalitica che accelera le reazioni chimiche necessarie per la polimerizzazione degli aminoacidi e contiene un "tunnel" dal quale fuoriesce la proteina nascente.
Figura 1 | Sintesi proteica
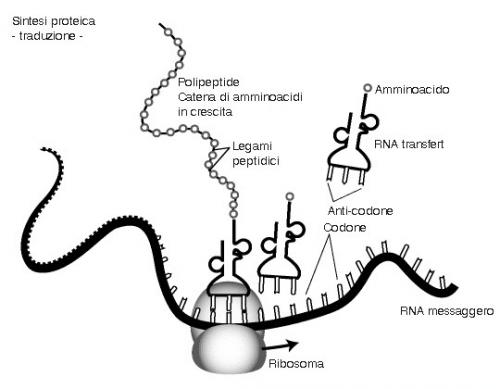
Schema semplificato del meccanismo di sintesi proteica, che mostra come i ribosomi decodificano il messaggio genetico allungando la catena polipeptidica un aminoacido dopo l’altro.
Il meccanismo generale della sintesi proteica era noto fin dalla fine degli anni 70, ma per capirne esattamente la dinamica era necessario decifrare a livello atomico la struttura dei ribosomi. Oltre all'immenso valore conoscitivo, una simile impresa aveva anche importanti risvolti pratici. I ribosomi, infatti, sono uno dei bersagli preferiti dagli antibiotici, sostanze prodotte da vari microorganismi, che uccidono le cellule o ne bloccano la crescita. Gli antibiotici che inibiscono la sintesi proteica si legano a regioni funzionalmente importanti dei ribosomi, interferendo con la loro attività. Gli antibiotici in uso come farmaci interagiscono esclusivamente con i ribosomi dei batteri patogeni e non con quelli dell'ospite umano, sfruttando sottili differenze che esistono fra i due tipi di particelle. Conoscere la struttura atomica dei ribosomi avrebbe permesso di capire come funzionano gli antibiotici e come essi discriminino fra ribosomi batterici ed eucariotici, consentendo di progettare in laboratorio nuove varianti di questi farmaci.
La mappatura atomica di strutture grandi e complesse come i ribosomi si presentava come un'impresa monumentale. In pratica, i ribosomi dovevano essere cristallizzati e poi analizzati mediante diffrazione dei raggi X, ottenendo un tracciato da cui ricavare la posizione dei singoli atomi (Figura 2). Questa procedura era stata applicata con successo a singole proteine, ma un ribosoma è circa 1000 volte più grande di una proteina media e contiene centinaia di migliaia di atomi. Il primo ostacolo era proprio ottenere cristalli ribosomici di buona qualità. I ribosomi più studiati, sui quali erano disponibili molti dati genetici e biochimici, erano quelli del batterio Escherichia coli (o E. coli). Ma nessuno era riuscito a cristallizzare i ribosomi di E.coli.
Figura 2 | La diffrazione dei raggi X 
La sorgente di raggi X (freccia spessa) viene creata utilizzando sincrotroni, tunnel circolari dove gli elettroni sono accelerati fino a velocità prossime a quella della luce. Quando i raggi X colpiscono il cristallo, vengono dispersi in varie direzioni, creando pattern caratteristici su uno schermo rivelatore. Analizzando questi pattern con appositi software, i ricercatori possono determinare la posizione di ciascun atomo nel cristallo.
La svolta ci fu quando Ada Yonath intuì che per avere buoni cristalli bisognava utilizzare ribosomi di organismi estremofili, cioè capaci di crescere in condizioni proibitive per cellule "normali". I candidati migliori erano batteri alofili, che vivono ad altissime concentrazioni saline, e termofili, capaci di adattarsi a temperature anche superiori a 100°C. Il razionale era che le macromolecole di queste cellule, ribosomi inclusi, dovevano essere piuttosto rigide, e quindi più propense a formare strutture cristalline ordinate.
La scommessa si rivelò vincente. All'inizio degli anni 80 Ada Yonath aveva buoni cristalli di entrambe le subunità ribosomiche e iniziò la raccolta dei dati. Alcuni problemi tecnici che ne ostacolavano l'interpretazione furono risolti grazie al contributo di Tom Steitz. Fu proprio Steitz a pubblicare, nel 1998, la prima struttura a bassa risoluzione (circa 9 Å) della subunità maggiore, nella quale non si distinguevano ancora i singoli atomi ma era chiaramente visibile il profilo degli rRNA. I progressi successivi furono molto rapidi: nel 2000, quasi contemporaneamente, Tom Steitz ottenne la struttura ad alta risoluzione (2.5 Å) della subunità maggiore mentre Ada Yonath e Venki Ramakrishnan pubblicarono quella della subunità minore (Figura 3). La mappatura a livello atomico delle funzioni ribosomiche era finalmente possibile. Grazie anche al contributo di molti altri gruppi di ricerca, che nel tempo hanno studiato vari aspetti della sintesi proteica in organismi diversi, oggi comprendiamo nei dettagli molecolari come il messaggio genetico proveniente dal DNA sia tradotto in proteine, cioè in funzioni specifiche delle cellule e degli organismi.
Figura 3 | truttura atomica ad alta risoluzione della subunità ribosomica minore (a sinistra) e di quella maggiore (a destra)
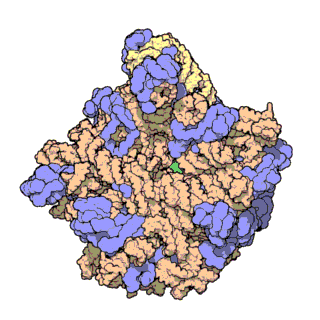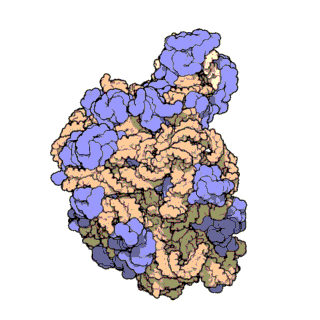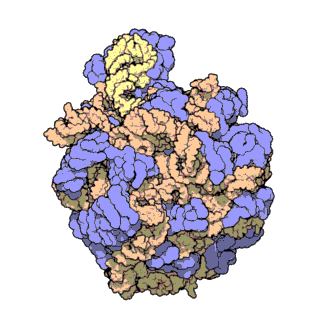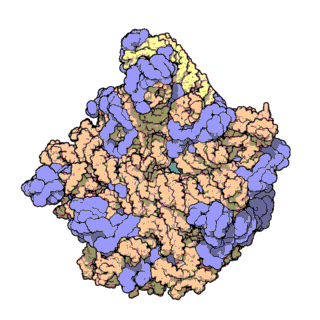
L’rRNA è colorato in rosa, le proteine in azzurro. Il punto verde al centro della subunità maggiore mostra la posizione del centro catalitico dove si formano i legami peptidici.
I tre vincitori del Nobel per la Chimica 2009 hanno, fra l'altro, ottenuto strutture che mostrano come gli antibiotici si leghino ai ribosomi e ne ostacolino le funzioni (Figura 4). Alcuni impediscono il distacco delle proteine nascenti; altri inibiscono la formazione del legame peptidico fra gli aminoacidi; altri ancora provocano errori nella traduzione del messaggio genetico. Questi dati vengono attualmente utilizzati da alcune industrie per sviluppare nuovi antibiotici, capaci di aggredire i sempre più diffusi batteri pluriresistenti a questi farmaci.
Figura 4 | La struttura atomica di un ribosoma batterico con un antibiotico legato
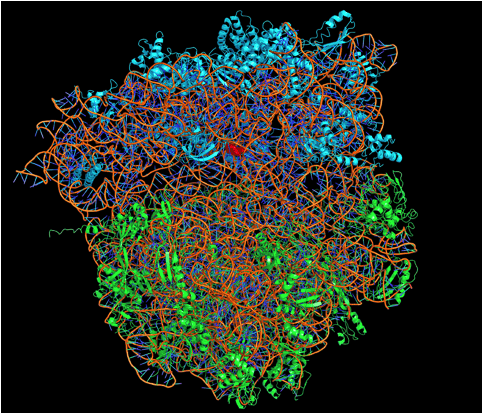
Le molecole di rRNA sono colorate in arancio, le proteine della subunità minore sono azzurre e quelle della subunità maggiore sono verdi. Una molecola di antibiotico, in rosso, è legata alla subunità minore.